Podręcznik
3. Sieci neuronowe głębokie
3.7. Sieć U-NET w segmentacji obrazów biomedycznych
Segmentacja obrazów biomedycznych ma za zadanie wyłowić w obrazie obszary należące do tej samej klasy (zbiór pikseli o podobnej jasności). Zadanie należy do bardzo trudnych ze względu na dużą różnorodność obszarów, zmienność położenia względnego pikseli względem siebie czy rozmycia granic obszarów jak również barw. Jednym ze skutecznych rozwiązań w tym zakresie jest sieć U-net stworzona przez autorów pracy [56]. Struktura tej sieci przypomina kształt litery U i zawiera podsieć CNN zwężającą oraz drugą podsieć CNN rozszerzającą, obie współpracujące ze sobą. Struktura takiej sieci przedstawiona jest na rys. 6.22. Na każdym poziomie przetwarzania stosowane są 2 warstwy konwolucyjne.
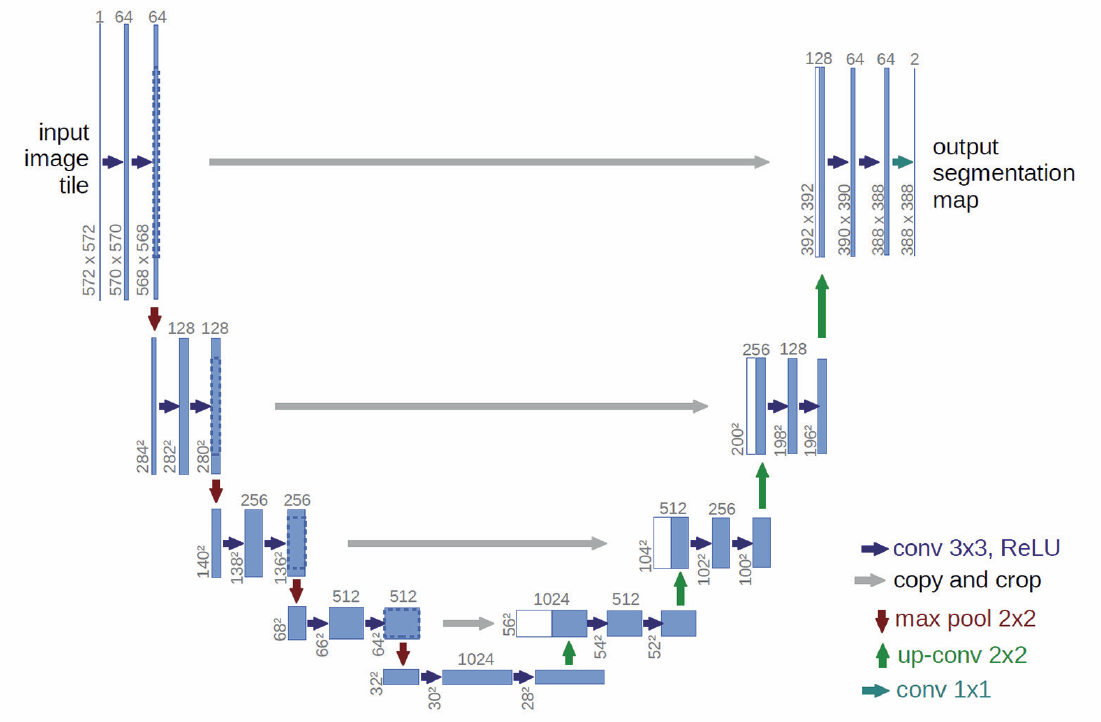
Obrazy występujące w warstwach zwężających (lewa część sieci) o coraz mniejszych wymiarach (operacje pooling) zmniejszają rozdzielczość wyników, co powoduje pogorszenie lokalizacji poszczególnych segmentów. Ta część sieci nazywana jest również enkoderem. W części rozszerzającej struktury (prawa część sieci) operacja pooling w każdej warstwie jest zastąpiona przez operację odwrotną (interpolację) zwaną up-sampling (pooling 2×2 zastępuje 4 piksele przez 1 piksel, natomiast up-sampling 2×2 działa odwrotnie: 1piksel jest replikowany w 4 piksele). Ta część sieci jest również nazywana dekoderem. Sieć U-net nie posiada warstw w pełni połączonych (fully connected layers) typowych w rozwiązaniach CNN.
Część zwężająca U-net stosuje konwolucję przy użyciu maski filtrującej 3×3 z krokiem stride=2, funkcję aktywacji ReLU i max pooling 2×2. W efekcie wymiar mapy cech (obrazu) w każdej kolejnej warstwie jest zmniejszany dwukrotnie, ale jednocześnie generowana jest liczba obrazów wzrastająca 2-krotnie.
Warstwy rozszerzające stosują operację interpolacji (up-sampling) o wymiarze 2×2, w której jeden piksel jest zastępowany 4 pikselami. Stosowane są różne metody, na przykład metoda najbliższych sąsiadów, zastosowanie splinów, itp. Za bardziej optymalną uważa się operację konwolucji transponowanej, zwanej również dekonwolucją, definiowanej jako operacja odwrotna do konwolucji (w praktyce określa się dekonwolucję stosując przekształcenie odwrotne Fouriera). Operacja 2×2 dekonwolucji zwiększa 2-krotnie wymiar mapy cech (obrazu wynikowego) przy dwukrotnie zmniejszonej ich liczbie. Jednocześnie na każdym poziomie warstwowym up-samplingu następuje dołączenie odpowiedniego obrazu niższej rozdzielczości z części zwężającej (część biała obrazów wejściowych na danym poziomie). Podobnie jak w warstwie zwężającej stosowana jest konwolucja z filtrem 3×3 i operacja ReLU. W części końcowej warstwy wyjściowej stosowana jest konwolucja 1×1 tworząca wysegmentowany obraz wyjściowy, w którym każdy piksel jest reprezentowany przez przypisany do niego numer klasy (podział na klastry reprezentujące oddzielne segmenty).
Uczenie sieci odbywa się z udziałem obrazów zawierających segmenty opisane przez eksperta. Proces uczenia ma za zadanie zminimalizować sumę różnic między stopniami szarości pikseli tworzących ROI segmentu zakreślonego przez eksperta i tych wygenerowanych przez warstwę końcową sieci U-net (funkcja celu z użyciem definicji kross-entropijnej). Odbywa się przy zastosowaniu metody stochastycznej największego spadku (SGD) z momentem rozpędowym (SGDM). Szczegóły można znaleźć w publikacji [56].
